library(caret)
library(kernlab)
data(spam)
noTreino = createDataPartition(y = spam$type, p = 0.75, list = F)
treino = spam[noTreino,]
teste = spam[-noTreino,]
# Para fazer a reamostragem por k-fold vamos utilizar o comando createFolds():
folds = createFolds(y = spam$type, k = 10, list = T, returnTrain = T)7 Cross Validation (Validação Cruzada)
Existem diversos métodos de aprendizado de máquina que podemos usar para construir um preditor. Então como saber qual método é melhor? Um jeito de fazer isso é usando a validação cruzada.
A Validação Cruzada nos permite comparar diferentes métodos de aprendizado de máquina ou parâmetros para o método escolhido e avaliar qual funcionará melhor na prática.
Então o que vamos fazer é, para cada método,
Separar os dados em conjunto de treino e conjunto de teste.
Treinar um modelo no conjunto de treino.
Avaliar no conjunto de teste
Repetir os passos 1-3 e estimar o erro.
Bem, já sabemos que não é uma boa ideia usar toda a base de dados para treinar o nosso preditor e então podemos dividir por exemplo os primeiros 75% dos dados para treino e 25% finais para teste. Mas, e se esse não for o melhor jeito de dividir nossos dados? E se o melhor jeito de fazer essa divisão for usando os primeiros 25% para teste e o restante para treino? A Validação cruzada leva em consideração todas essas divisões usando uma de cada vez e tirando a média dos resultados no final. Para isso veremos como realizar alguns métodos de reamostragem, para utilizarmos várias amostras possíveis e não ficarmos dependentes de uma única amostra.
7.1 Alguns Métodos de Reamostragem
Agora vamos compreender como fatiar os dados para realizarmos a reamostragem. Existem vários métodos possíveis mas vamos nos focar em três: k-fold, repeated k-fold e bootstrap.
7.1.1 K-fold
Este método consiste em fatiar os dados em k pedaços iguais. Utilizamos um pedaço para o teste e os demais para o treino. Então realizamos esse procedimento k vezes, de modo que em cada repetição um novo pedaço seja utilizado para o teste. Para avaliar o erro nós tiramos a média de todos os erros de todas as replicações.
Exemplo: K-fold com 10 partes:
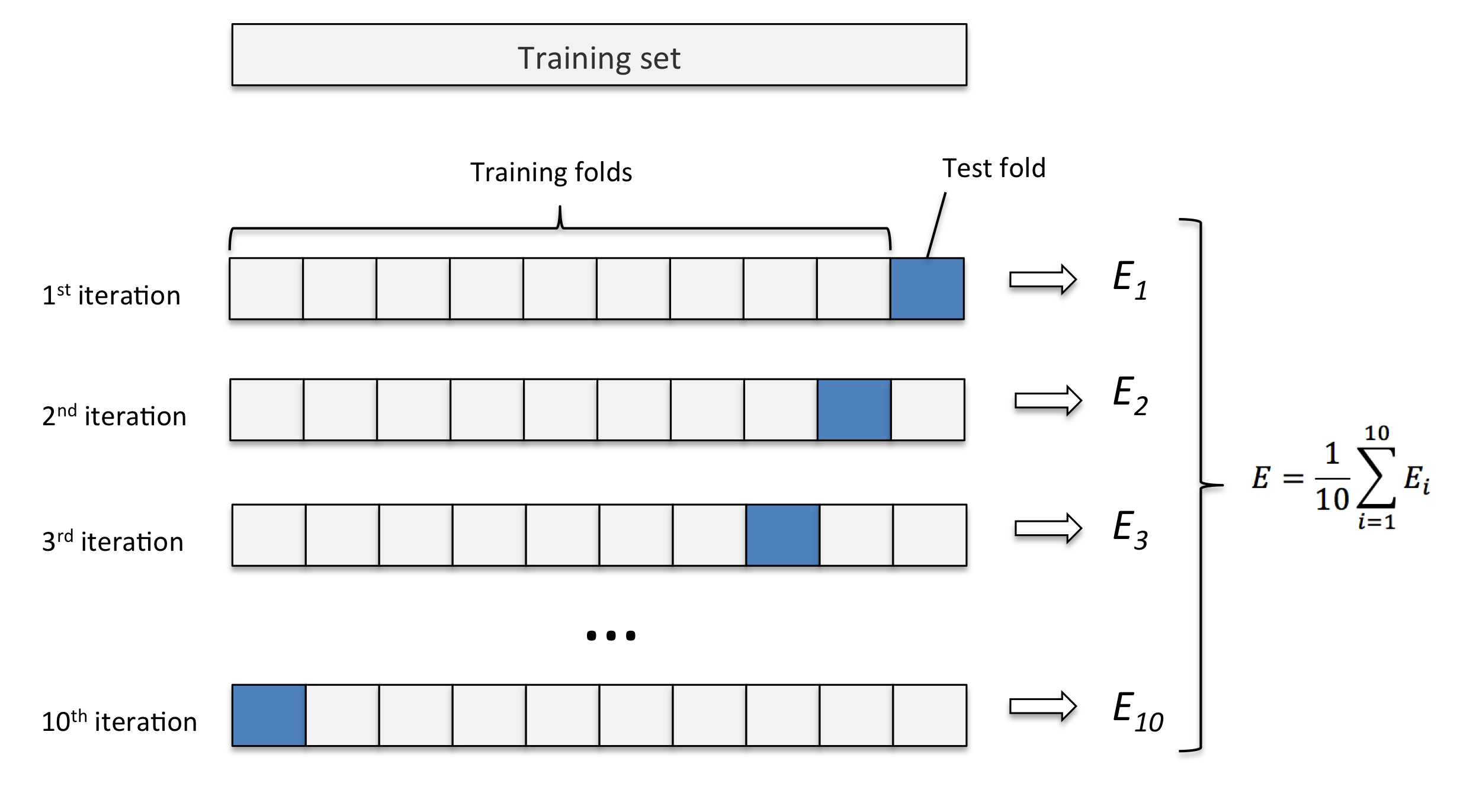
Quanto maior o k escolhido obtemos menos viés, porém mais variância. Em outras palavras, você terá uma estimativa muito precisa do viés entre os valores previstos e os valores verdadeiros, porém altamente variável. Agora quanto menor o k escolhido, mais viés e menos variância. Ou seja, não iremos necessariamente obter uma boa estimativa do viés, mas ela será menos variável.
OBS: Quando o k é igual ao tamanho da amostra, o método é também conhecido como leave-one-out.
Ex.: vamos utilizar reamostragem por k-fold no conjunto de dados spam.
Os principais argumentos da função createFolds() são:
y = a variável de interesse (no nosso caso, o tipo do e-mail);
k = o número (inteiro) de partições que você deseja.
list = argumento do tipo logical. Se TRUE → os resultados serão mostrados em uma lista, se FALSE → os resultados serão mostrados em uma matriz.
returnTrain = argumento do tipo logical. Se TRUE, retorna amostras treino. Se FALSE, retorna amostras teste.
Vamos verificar o tamanho de cada partição da nossa amostra treino:
sapply(folds,length)Fold01 Fold02 Fold03 Fold04 Fold05 Fold06 Fold07 Fold08 Fold09 Fold10
4140 4141 4141 4141 4141 4140 4142 4141 4141 4141 Agora vamos fazer o mesmo para a amostra teste:
folds = createFolds(y = spam$type, k = 10, list = T, returnTrain = F)
sapply(folds,length)Fold01 Fold02 Fold03 Fold04 Fold05 Fold06 Fold07 Fold08 Fold09 Fold10
460 460 460 461 460 461 460 460 460 459 Outra opção de realizar a reamostragem por k-fold é aplicá-la diretamente na função train.
controle = trainControl(method = "cv", number = 10)
modelo = caret::train(type ~ ., data = spam, method = "glm", trControl = controle)7.1.2 Repeated K-fold
O repeated k-fold se resume a repetir o método k-fold várias vezes, com o objetivo de melhorar nossa reamostragem.
Ex.: Vamos aplicar um método de treino 3 vezes em 10 folds.
controle = trainControl(method = "repeatedcv", number = 10, repeats = 3)
modelo = caret::train(type ~ ., data = spam, method = "glm", trControl = controle)7.1.3 Bootstrap
O bootstrap é uma técnica de reamostragem com o propósito de reduzir desvios e realizar amostragem dos dados de treino com repetições. Já vimos anteriormente que este é o método default do comando train(), onde é feito 25 reamostragens por bootstrap.
Embora esse seja o padrão podemos alterar através do comando trainControl(). Por exemplo, vamos alterar o número de reamostragens de 25 para 10.
controle = trainControl(method = "boot", number = 10)
modelo = train(type ~ ., data = spam, method = "glm", trControl = controle)Podemos também realizarmos bootstrap fora da função train(), utilizando o comando createResample().
folds = createResample(y = spam$type, times = 10, list = F)